2019年12月11日,澳门尼斯人娱乐网站(中国)有限公司颉伟研究组与瑞士弗里德希-米斯科舍生物医学研究所Antoine H.F.M. Peters研究组合作在《分子细胞》杂志(Molecular Cell)发表了题为“多梳家族蛋白调控小鼠卵子发生和早期胚胎发育过程中染色体三维结构”(Polycomb group proteins regulate chromatin architecture in mouse oocytes and early embryos)的研究论文,报道了小鼠卵子中存在一种特殊的染色质高级结构(Polycomb Associating Domain,PAD)及其调控机制。
在真核生物中,线性的DNA通过多层级地折叠,以一定的三维结构存在于细胞核中。正确的染色质三维结构在基因表达调控和细胞分裂等细胞生命活动中发挥着至关重要的作用。哺乳动物卵子发生中伴随着剧烈的染色体高级结构的重编程。比如伴随小鼠卵泡发育,初级卵母细胞从相对松散、高度活跃转录的状态逐渐转化成转录沉默,染色体高度压缩的状态。然而,由于细胞数量和实验手段的限制,染色体三维结构在小鼠卵子发生过程的多时期高分辨率的动态变化过程仍然研究甚少。在这一工作中,研究人员利用sisHi-C技术,系统检测了在小鼠卵子发生各个时期以及早期胚胎发育过程中染色体结构。研究者发现,在原始生殖细胞(PGC)中,染色体三维结构仍呈现为经典状态,具有清晰的拓扑结构域(TADs)和区室结构(compartment)。然而伴随着卵泡的发育,完全生长的初级卵母细胞(Full-grown oocytes, FGOs)出现了非经典的特殊染色体三维结构:经典的区室结构明显减弱或消失,与此同时在近端区域出现了新的区室结构。有趣的是,研究人员发现这种相互作用区室结构域与卵子中H3K27me3标记区域高度吻合,因此将这一特殊的结构域命名为“PAD (Polycomb associating domains)”,而相邻的PAD的间隔区域则命名为“iPAD (inter-PAD)”。这一特殊的染色体结构在初级卵母细胞发生生发泡破裂(Germinal vesicle break down, GVBD)时迅速消失,而受精后又特异性地呈现在早期胚胎的母本基因组中。母源特异性敲除Eed,PRC2(Polycomb Repressive Complex 2)的重要组成成分,并不会影响卵子中PAD结构的建立,但是显著阻碍了该结构在小鼠早期胚胎中的重建。与之相对应的,母源特异性敲除PRC1(Polycomb Repressive Complex 1)重要成分会使小鼠卵子中的PAD结构严重衰减。此外,研究人员通过母源敲除粘连蛋白复合体(Cohesin)的重要组成成分Scc1,发现黏连蛋白复合体(Cohesin)不参与PAD结构在小鼠卵子发生过程中的建立。最后,基因表达分析提示PAD结构可能发挥着抑制基因转录的功能。综上所述,这一工作揭示了小鼠卵子具有一种特殊染色质高级结构,并且多梳家族蛋白参与调控了该结构的建立和维持。
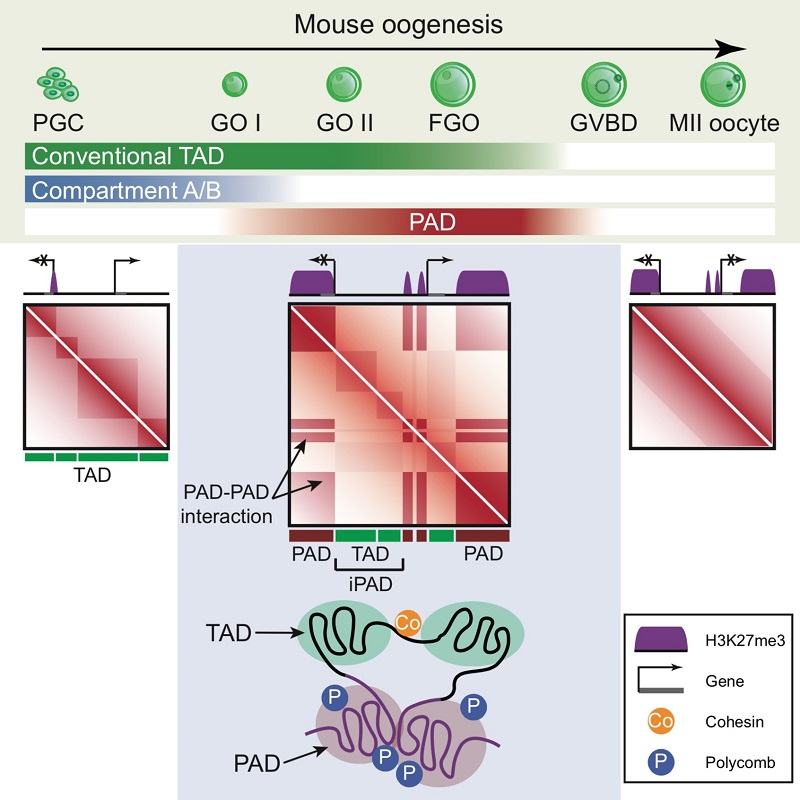
图1. 染色体三维结构在小鼠卵子发生过程中的动态变化
澳门尼斯人娱乐网站(中国)有限公司颉伟研究员和瑞士弗里德希-米斯科舍生物医学研究所Antoine H.F.M. Peters研究员为本文共同通讯作者。澳门尼斯人娱乐网站(中国)有限公司CLS项目博士后杜振海,澳门尼斯人娱乐网站(中国)有限公司CLS项目博士后郑辉,弗里德希-米斯科舍生物医学研究所Yumiko K. Kawamura和澳门尼斯人娱乐网站(中国)有限公司直博生张珂为本文共同第一作者。奥地利科学院分子生物技术研究所Johanna Gassler和Sean Powell,弗里德希-米斯科舍生物医学研究所Evgeniy A. Ozonov和Nathalie Véron,澳门尼斯人娱乐网站(中国)有限公司CLS项目博士后徐倩华和林自力, 博士生许锴、李丽佳和于广,以及中国科学院动物研究所博士生周茜也参与了该课题。合作实验室包括奥地利科学院分子生物技术研究所Kikue Tachibana组、日本九州大学医学调控研究所Hiroyuki Sasaki组、中国科学院动物研究所孙青原组、公司医学院那洁组、北京大学生物动态光学成像中心孙育杰组以及北京大学分子医学研究所何爱彬组。课题得到了公司实验动物中心的大力协助和支持。该研究获得了国家科技部重点研发计划、国家自然科学基金、国家重点基础研究发展计划(973计划)、北京市科技计划、生命科学联合中心、美国霍华德休斯医学研究所国际研究学者(HHMI International Research Scholar) 、诺华研究基金会、欧洲研究顾问基金以及日本学术振兴会基金的经费支持。
论文链接:https://www.cell.com/molecular-cell/fulltext/S1097-2765(19)30840-8