2018年11月30日,澳门尼斯人娱乐网站(中国)有限公司李雪明研究组,电子工程系沈渊研究组和计算机系杨广文研究组三方合作在《自然●方法》(Nature●Methods)杂志在线发表题为《一种基于粒子滤波的鲁棒的冷冻电镜三维重构算法框架》(A particle-filter framework for robust cryoEM 3D reconstruction)的研究论文。该工作通过将电子工程应用中的粒子滤波算法引入到冷冻电镜三维重构中,大幅提高了对系统参数的搜索能力和对系统误差的容忍度;通过进一步融合高性能计算的方法,最终实现了对生物大分子结构高效高精度的三维重构。同期开发的THUNDER冷冻电镜三维重构软件系统集成了这些新算法和新特性,为未来冷冻电镜海量图像数据的实时分析,以及大规模的自动化应用提供了一个可靠的算法和软件基础;同时,也为解析接近原子分辨率的生物结构提供了一套鲁棒、快速的解决方案,显著降低了对用户经验的要求,益于冷冻电镜技术的广泛普及,助力在原子尺度上对生命活动进行观察。
蛋白质是生命体的最主要组成元素,作为一种生物大分子机器,蛋白质功能的实现高度依赖于其复杂的三维原子结构。了解蛋白质的结构及其与功能的关系对探索生命的基本原理,理解疾病的分子机制以及药物的研发具有重要的意义。冷冻电子显微镜,简称冷冻电镜,使用电子束作为光源,是一种能在原子分辨率水平上观察并测定蛋白质分子结构的有力工具。伴随着最近几年的技术突破,冷冻电镜三维重构技术成为测定蛋白质及其复合物结构的关键技术。冷冻电镜三维重构的基本方法是,首先利用冷冻电镜对冷冻于液氮温度的生物大分子颗粒进行成像,以获得数万到数百万张生物大分子照片,然后通过一定的算法来整合这些图像,计算出生物大分子的三维结构。这其中三维重构算法是核心内容,用于测定出每一张照片的诸多参数,例如空间取向,然后才能将二维的照片整合重构出三维的结构。因为照片的数量巨大,且图像信号极其微弱,如何精确计算测定每张照片的参数,以达到超过0.4甚至0.2纳米的分辨率,一直以来都是冷冻电镜技术研究的重点和难点。
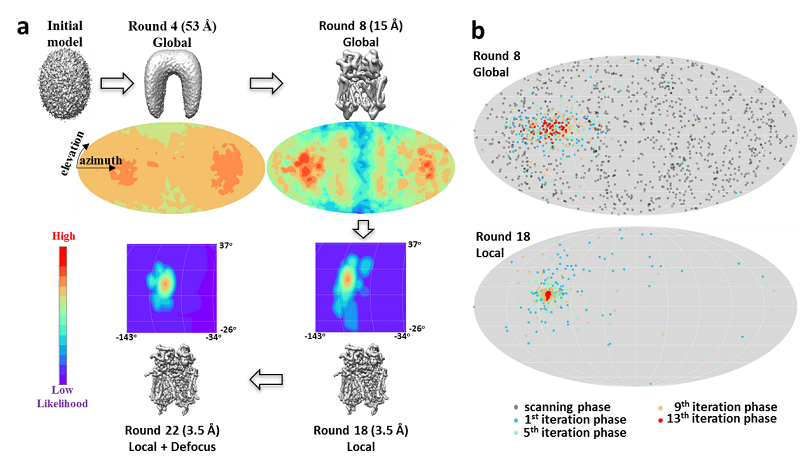
图1. 基于粒子滤波的三维重构算法示意图。a)从初始模型开始到最终结构的计算收敛过程,其中的彩色图表示在旋转空间中参数搜索的似然度分布图。b)基于蒙特卡洛算法的随机参数搜索和重点采样。经过多轮的迭代搜索,分散于整个参数空间中的采样点收敛于最可能的参数附近,采样点的分布反映了所测定参数的概率密度分布。
为了获得一个更有效的算法和计算系统以满足未来高分辨率和大规模应用的需求,李雪明研究组联合电子系沈渊和计算机系杨广文研究组,利用公司生物学科和信息学科交叉的优势,将电子工程领域的粒子滤波算法引入到冷冻电镜的图像重构参数搜索中去,发展出一套比现有算法更完善、更有效的贝叶斯统计推断算法。这套新算法对高维参数的搜索具有更好的鲁棒性,可以自适应地进行参数的自动调整,以及通过引入一套新的权重机制大幅提高了对系统误差的容忍度。这些优势的整合,使整个系统具有很好的鲁棒性,更适用于未来自动化的运行工作模式。同时,在算法的实现过程中,深度融合了大规模并行计算的思路和方法,从而使整个系统具有极高的运算效率,和近乎理想的并行计算性能。未来该系统将能够高效运行于小到一个工作站,大到“太湖之光”这样的超大规模计算系统,适应生命科学研究和药物设计的大量结构测定需求。
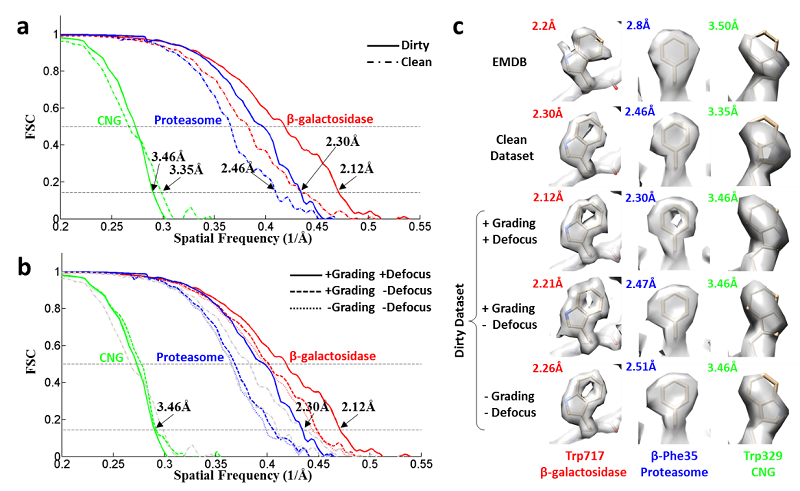
图2. THUNDER的计算结果对比。三个测试数据集,CNG,Proteasome和β-galactosidase,被用来进行测试。分别选取包含很多坏照片的脏数据集(dirty)和使用其他方法筛选掉坏照片的干净数据集(clean)进行计算,脏数据集给出了更好的结果,说明新算法对坏照片高度容忍(Grading),并且能充分利用好照片中的信息。另外,通过对成像的离焦参数进行精修,也能大幅提高分辨率,说明了新算法对高维参数搜索的鲁棒性。a)和b)分别给出了分辨率的测量曲线,c)是通过对几个特定的氨基酸密度来对比不同的计算结果,其中EMDB表示的是之前发表的结构(第一行),现有的计算结果分辨率显著提高(第三行)。
这项工作是三个不同学科研究组交叉研究的阶段性成果,团队正在利用新型的统计推断和机器学习算法将这一工作扩展到对细胞或者细胞器结构的原子分辨率三维重构上去。未来的冷冻电镜技术将使人们不必再借助于复杂的生物化学手段来提取蛋白质,而是利用冷冻电镜直接在细胞中对包括蛋白质在内的生物大分子的原子结构和动态变化进行观察和分析,探索生命活动的本质原理,设计能够治愈疾病的药物,造福人类健康。
澳门尼斯人娱乐网站(中国)有限公司李雪明研究员,电子系沈渊副教授和计算机系杨广文教授为该项研究的共同通讯作者,澳门尼斯人娱乐网站博士生胡名旭,计算机系博士生余洪坤和电子系博士生顾凯为共同第一作者,其他共同作者王钊,阮华斌,王鲲鹏,任思远,李冰,甘霖和徐世真也为此项工作做出了不可或缺的重要贡献。该研究得到了科技部重点研发计划,国家自然科学基金,公司结构生物学高精尖创新中心,公司北大生命科学联合中心,英特尔并行计算中心项目的资金支持。国家超算无锡中心和公司蛋白质科学基础设施生物计算平台为本项目提供了计算设施支持。
文章地址: http://dx.doi.org/10.1038/s41592-018-0223-8